泛基因组
泛基因组是为描述一個物種基因组而提出的概念,指同一细菌物种中所有菌株中所有基因的集合,而不单纯以某个菌株为一个物种的全基因组。會需要使用泛基因組的原因是水平基因轉移造成不同菌株之間所擁有的基因相差甚大。
泛基因组的英文是Pan-genome, Pan- 来自希腊语词汇 παν,意思是「全部的」[1]。在1987年[2],細菌物种的概念是大于70%的DNA相关联,并具有相同的表型性。但是随着对大量细菌基因组测序的完成,这种概念区分方法变得模糊、不确定——测定同一物种的不同菌株,常会有新的基因出现。這個概念常被用於總體基因體學、演化生物學,應用的物種也有被延伸到植物或古菌。
泛基因組包含核心基因和附加基因。核心基因是所有個體都擁有的基因,附加基因則為非核心基因的基因[3][4]。在附加基因組中,若僅有一個個體具有該基因,則可稱之為獨特基因(英語:)。為了允許註解及基因序列組裝的錯誤,對核心基因組較為寬鬆的定義可稱之為軟核心基因(soft core gene),其定義為於 95%以上的個體具有此基因。[5]
各物種有兩種泛基因組,分別為開放型泛基因組和閉合型泛基因組。閉合型泛基因組的物種隨著被定序的個體數量增加,泛基因組的大小增加有限,使得完整的泛基因組大小是在數學上可以預測的。相反地,開放型基因組的物種隨著定序的個體增加,其泛基因組的大小並未收斂。影響物種是為何種泛基因組,受到族群大小、水平基因轉移等影響。[4] [6]
範例
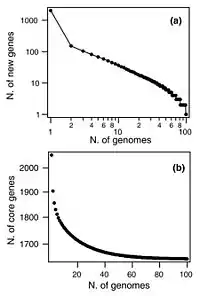
最早的泛基因組概念是由Tettelin等人在研究六株B型鏈球菌的菌株時提出的 [8],最主要的概念就是找出所有菌株共有的核心基因(約佔每隻菌株基因體的80%),以及非必須的附加基因(有可能只存在於一小部分的菌株中,或甚至只出現在單一菌株的基因體中),並透過核心基因與附加基因的組合來描述B型鏈球菌。外推法模型預測這隻細菌的基因庫會持續增長,且即使在加入數百隻細菌後,新的基因將會不斷加入,顯示這隻細菌的總基因庫相當龐大。[8]
44株肺炎鏈球菌菌株的泛基因體分析與B型鏈球菌有著不同的特徵形態—雖然每這44株細菌的基因體都會貢獻新的基因給肺炎鏈球菌的基因庫,但是基因增長速度會隨著細菌數量的增加而明顯變慢。事實上,外推法預測在50隻細菌加入後,新基因的數量將會降到零。顯而易見的是這個特徵並非所有細菌共有的。對肺炎鏈球菌來說,新基因最主要的來源是和緩鏈球菌的水平基因轉移。肺炎鏈球菌的泛基因大小將會與菌株數量成對數級增加,而與新加入基因體的核苷酸多形位點數量形成線性關係,顯示年紀越大的菌株將會越有可能得到新基因。[7]
最後一個例子是原綠球藻的核心基因體與完整的泛基因體大小比對。[9] 在這個例子中,核心基因體明顯地比泛基因體小很多,而不同生態型的原綠球藻會有著不同的基因組合。另一篇2015年的研究也在從人類身上不同部位分離普雷沃氏菌屬,並重建泛基因體後,發現這隻細菌有著龐大基因庫,且屬於開放式泛基因組類型。[10]
軟體
隨著越來越多人關注泛基因體學,有更多的軟體可以用來建立與分析泛基因體、繪圖、註解。[11] [11]
2014 年底最高引用數的的軟體為 [11]Panseq[12] 以及 pan-genome analysis pipeline(PGAP)[13]。其他的選擇包含 BPGA - A Pan-Genome Analysis Pipepline for prokaryotic genomes、GET_HOMOLOGUES、Roary 與 CD-HIT[14] GET_HOMOLOGUES [15] or Roary.[16] 。針對各家軟體的比較,可以參照 2015 年的一篇文章。[11]
針對植物泛基因體的軟體有 GET-HOMOLOGUES-EST[17] 。有關植物泛基因體可以參照 2015 的一篇文章[18]。
参考资料
- http://www.pnas.org/content/102/39/13950.long
- http://ijsb.sgmjournals.org/content/37/4/463.short%5B%5D
- Medini, Duccio; Donati, Claudio; Tettelin, Hervé; Masignani, Vega; Rappuoli, Rino. . Current Opinion in Genetics & Development. 2005, 15 (6): 589–594. PMID 16185861. doi:10.1016/j.gde.2005.09.006.
- Vernikos, George; Medini, Duccio; Riley, David R; Tettelin, Hervé. . Current Opinion in Microbiology. 2015, 23: 148–154. doi:10.1016/j.mib.2014.11.016.
- Kaas RS, Friis C, Ussery DW, Aarestrup FM. . BMC Genomics. October 2012, 13: 577. PMC 3575317. PMID 23114024. doi:10.1186/1471-2164-13-577.
- McInerney, James O.; McNally, Alan; O'Connell, Mary J. . Nature Microbiology. 2017-03-28, 2 (4). ISSN 2058-5276. doi:10.1038/nmicrobiol.2017.40 (英语).
- Donati, C; Hiller, N. L.; Tettelin, H; Muzzi, A; Croucher, N. J.; Angiuoli, S. V.; Oggioni, M; Dunning Hotopp, J. C.; Hu, F. Z.; Riley, D. R.; Covacci, A; Mitchell, T. J.; Bentley, S. D.; Kilian, M; Ehrlich, G. D.; Rappuoli, R; Moxon, E. R.; Masignani, V. . Genome Biology. 2010, 11 (10): R107. PMC 3218663. PMID 21034474. doi:10.1186/gb-2010-11-10-r107.
- Tettelin, H; Masignani, V; Cieslewicz, M. J.; Donati, C; Medini, D; Ward, N. L.; Angiuoli, S. V.; Crabtree, J; Jones, A. L.; Durkin, A. S.; Deboy, R. T.; Davidsen, T. M.; Mora, M; Scarselli, M; Margarit y Ros, I; Peterson, J. D.; Hauser, C. R.; Sundaram, J. P.; Nelson, W. C.; Madupu, R; Brinkac, L. M.; Dodson, R. J.; Rosovitz, M. J.; Sullivan, S. A.; Daugherty, S. C.; Haft, D. H.; Selengut, J; Gwinn, M. L.; Zhou, L; 等. . Proceedings of the National Academy of Sciences. 2005, 102 (39): 13950–5. PMC 1216834. PMID 16172379. doi:10.1073/pnas.0506758102.
- Kettler GC, Martiny AC, Huang K, Zucker J, Coleman ML, Rodrigue S, Chen F, Lapidus A, Ferriera S, Johnson J, Steglich C, Church GM, Richardson P, Chisholm SW. . PLoS Genetics. 2007, 3 (12): e231. ISSN 1553-7390. PMC 2151091. PMID 18159947. doi:10.1371/journal.pgen.0030231.
- Gupta VK, Chaudhari NM, Dutta C. . BMC Genomics. 2015, 16 (153). PMC 4359502. PMID 25887946. doi:10.1186/s12864-015-1350-6.
- Xiao, Jingfa; Zhang, Zhewen; Wu, Jiayan; Yu, Jun. . Genomics, Proteomics & Bioinformatics. 23 February 2015, 13 (1): 73–76 [2017-01-28]. doi:10.1016/j.gpb.2015.01.007.
- Laing, Chad; Buchanan, Cody; Taboada, Eduardo; Zhang, Yongxiang; Kropinski, Andrew; Villegas, Andrea; Thomas, James; Gannon, Victor. . BMC Bioinformatics. 15 September 2010, 11 (1): 461 [2017-01-28]. doi:10.1186/1471-2105-11-461.
- Zhao, Yongbing; Wu, Jiayan; Yang, Junhui; Sun, Shixiang; Xiao, Jingfa; Yu, Jun. . Bioinformatics. 29 November 2011, 28 (3): 416–418 [2017-01-28]. PMC 3268234. doi:10.1093/bioinformatics/btr655.
- Chaudhari NM, Gupta VK, Dutta C. . Scientific Reports. 2016, 6 (24373). PMC 4829868. PMID 27071527. doi:10.1038/srep24373.
- Contreras-Moreira B, Vinuesa P. (PDF). Appl Environ Microbiol. October 2013, 79 (24): 7696–701. PMC 3837814. PMID 24096415. doi:10.1128/AEM.02411-13.
- Page AJ, Cummins CA, Hunt M, Wong VK, Reuter S, Holden MT, Fookes M, Falush D, Keane JA, Parkhill J. . Bioinformatics. July 2015, 31 (22): 3691–3693. PMC 4817141. PMID 26198102. doi:10.1093/bioinformatics/btv421.
- Contreras-Moreira B, Cantalapiedra CP, García-Pereira MJ, Gordon SP, Vogel JP, Igartua E, Casas AM, Vinuesa P. . Front. Plant Sci. February 2017, 8: 184. PMC 5306281. PMID 28261241. doi:10.3389/fpls.2017.00184.
- Golicz AA, Batley J, Edwards D. . Plant Biotechnol J. November 2015, 14 (4): 1099–1105. PMID 26593040. doi:10.1111/pbi.12499.